Checkm
lineage-specific(世系特异性)==【推荐方法】==
lineage-specific(世系特异性)(使用该方法需下载checkM的参考数据集,并设置安装在相应位置)
##下载数据库,并解压设置路径
wget -c https://data.ace.uq.edu.au/public/CheckM_databases/checkm_data_2015_01_16.tar.gz
tar -zxvf checkm_data_2015_01_16.tar.gz
checkm data setRoot $PATH/checkm_data#####我的指令
checkm lineage_wf -t 46 -x fa --nt --tab_table -f /home/chenl/RA/BGI/assembly/RAH26A/bin_qa_result/RAH26A_bins_qa.txt /home/chenl/RA/BGI/assembly/RAH26A/bin /home/chenl/RA/BGI/assembly/RAH26A/bin_qa_result(题外话:中间会用到pplacer,这一步也可以设置线程数,默认是1)
结果
得到bins_qa.txt
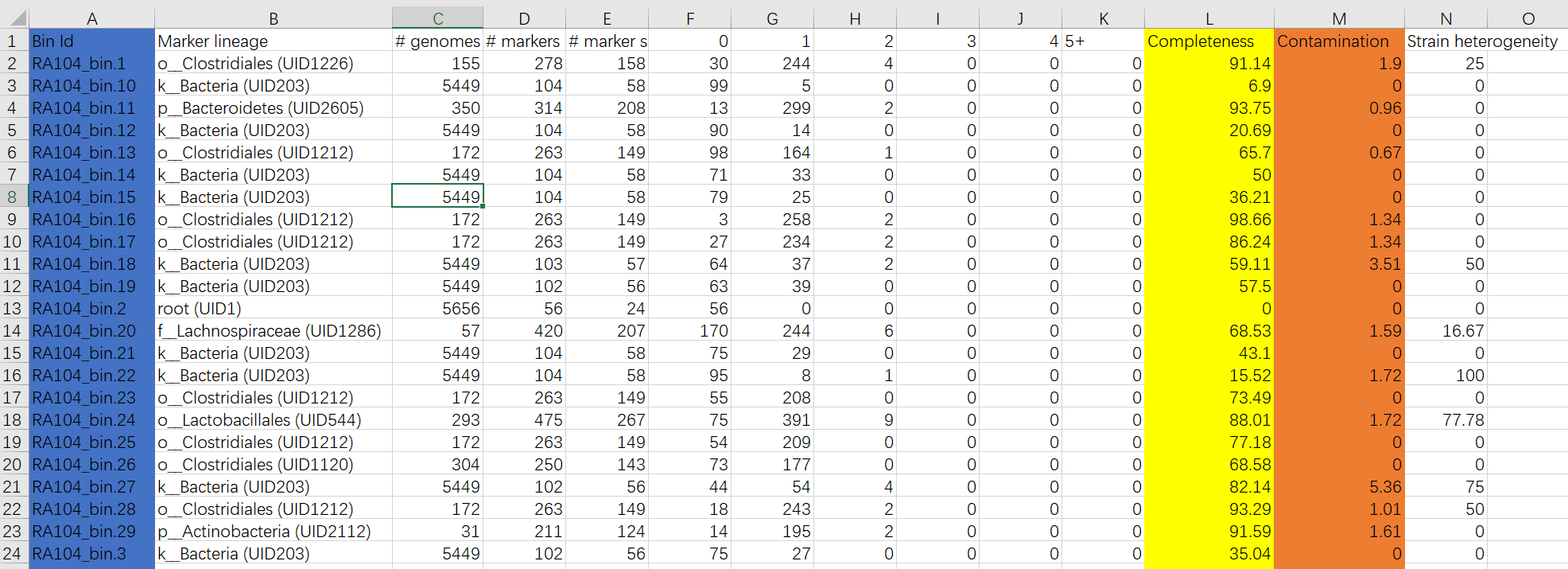 关键点:BIn Id(唯一的)、completeness(完整度)、Contamination(污染度)
关键点:BIn Id(唯一的)、completeness(完整度)、Contamination(污染度)
得到的结果是一个样本的所有bin,现在需要分析所有样本,就需要把所有样本整合到一起,那就是Python脚本,我自己上网随便搜的一个。
整合之后就是一个含有所有bins的污染度和完整度信息,就可以画图了。

